- PrognoScan A new database for meta-analysis of the prognostic value of genes.
水野英明1,2、北田邦雄1、中井謙太3、皿井明倫2
1中外製薬、2九州工業大学、3東京大学
目下、様々な遺伝子について癌との潜在的な関連が報告されている。こうした遺伝子を評価する主要な方法の一つは患者予後との関連を調べることである。近年、臨床情報の付随した癌マイクロアレイデータが大量に公開され、遺伝子発現と患者予後とを結びつける機会が提供されている。しかしながら、効率的なデータ解析のためのプラットフォームの開発が遅れており、利用は進んでいない。
遺伝子発現のような連続的な測定値を元に生存解析を行う場合、患者を発現の高い順に2等分、3等分といった具合に便宜的に群分けをし、群間のリスクを比較する方法がよく用いられる。しかし、これは必ずしも生物学的意味を反映するものではない。従って、仮説をあらかじめ持たない場合、適切な閾値を決めることが解析上の困難の一つとなる。ITTACAやREMBRANDTといった癌マイクロアレイデータの先駆的データベースでの解析が効率的に行えないのはこのためである。
Minimum P-value approachは連続的な測定値の中から、リスク分離を行う最適な閾値を見つけるための網羅的な手法であり、これまで腫瘍サイズ、細胞周期指標、遺伝子コピー数などの解析で有効性が示されている。最近、BUB1、HOXB4、MYCといった遺伝子について、癌へ寄与する発現量の閾値が報告され、この手法を癌マイクロアレイデータへ適用する妥当性が示された。こうした状況を踏まえ、我々は、1)臨床情報の付随した癌マイクロアレイデータの網羅的なコレクション、2)minimum P-value approachを用いた遺伝子発現に基づく生存解析ツール、の2つの特徴を持つデータベース”PrognoScan”を開発した。このデータベースは遺伝子発現−癌患者予後の関連の体系的な横断検索を可能にし、癌研究を加速させることが期待される。
PrognoScanはhttp://www.prognoscan.org/で公開されている。
- Screenshots
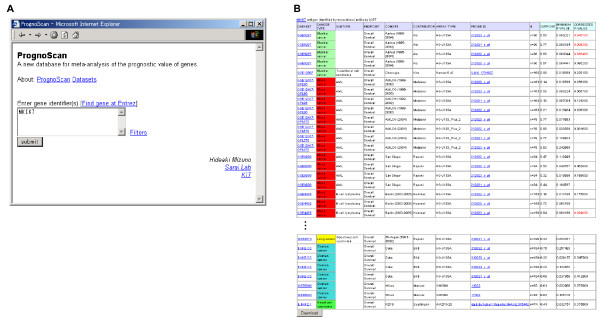
PrognoScan画面その1。(A)トップページは簡素で、遺伝子ID を入力するだけで使用できる。(B)サマリテーブル。列はデータセット・癌種・サブタイプ・エンドポイント・コホート・データ取得者・プラットフォーム・プローブID・患者数・最適分割点・Pmin・Pcorから成る。5%水準で有意なPcorは赤く強調される。各データセットには元データがアーカイブされてあるパブリック・ドメインへのリンクがついている。サマリテーブル内のプローブIDをクリックすることで各テストの詳細なレポートを見ることができる。下部にあるボタンを使ってテーブルをタブ区切りファイルでダウンロードすることができる。
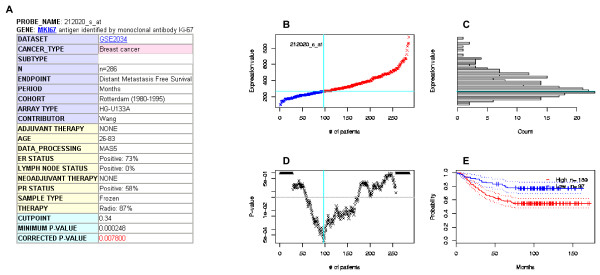
PrognoScan画面その2。(A)アノテーションテーブル。治療歴・サンプル種類・臨床パラメータなどの詳細を表示する。(B)遺伝子発現プロット。遺伝子発現量順に患者が並びかえられている。X軸は累積の患者数、Y軸は遺伝子発現量を表す。水色の直線は患者を高発現群(赤)および低発現群(青)に二分するのに最適な分割点を示す。(C)遺伝子発現ヒストグラム。X軸に患者数、Y軸に遺伝子発現プロットと同じスケールで分布を表示する。最適分割点が水色の直線で示される。(D)P-valueプロット。各分割点において患者が二分され、高発現群と低発現群の生存率の違いがLog-rankテストによって検定される。X軸は発現プロットと同様に患者の累積数を表し、Y軸は補正前のP-valueを対数スケールで表す。P値を最小にする点は水色の直線で示される。灰色の直線は5%有意水準を示す。(E)Kaplan-meier生存曲線。最適分割点で二分された高発現群(赤)および低発現群(青)の生存曲線が示される。X軸は時間、Y軸は生存率を表す。各群の95%信頼区間が点線で示される。
- Links
統合ホームページ

